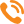![]() Медоносная пчела (Apis mellifera L.) является наиболее распространенным представителем рода Apis, обитающим на территории России. С использованием многокомпонентного анализа морфометрических признаков ее подразделяют на четыре эволюционные ветви [7]: A (Южная и Центральная Африка), M (Северная Африка и Западная Европа), C (север Средиземноморья, Центральная и Восточная Европа) и O (Средиземноморье, Средний и Ближний Восток), объединяющие 26 подвидов (Ruttner, 1992; Sheppardetal., 1997; Sheppard, Meixner, 2003; Meixneretal., 2011), а по некоторым данным — 28 подвидов (пород) (Engel, 1999). Некоторые из них, например A. m. mellifera, по результатам морфометрического анализа (Cornuetetal., 1975, 1978) подразделяют на дифференцированные локальные популяции. Российские и украинские ученые в качестве самостоятельного подвида выделяют карпатскую пчелу (A. m. carpatica), хотя зарубежные ученые считают ее ветвью краинской пчелы (A. m. carnica)[3].
Медоносная пчела (Apis mellifera L.) является наиболее распространенным представителем рода Apis, обитающим на территории России. С использованием многокомпонентного анализа морфометрических признаков ее подразделяют на четыре эволюционные ветви [7]: A (Южная и Центральная Африка), M (Северная Африка и Западная Европа), C (север Средиземноморья, Центральная и Восточная Европа) и O (Средиземноморье, Средний и Ближний Восток), объединяющие 26 подвидов (Ruttner, 1992; Sheppardetal., 1997; Sheppard, Meixner, 2003; Meixneretal., 2011), а по некоторым данным — 28 подвидов (пород) (Engel, 1999). Некоторые из них, например A. m. mellifera, по результатам морфометрического анализа (Cornuetetal., 1975, 1978) подразделяют на дифференцированные локальные популяции. Российские и украинские ученые в качестве самостоятельного подвида выделяют карпатскую пчелу (A. m. carpatica), хотя зарубежные ученые считают ее ветвью краинской пчелы (A. m. carnica)[3].
Развитие ДНК-технологий открыло новые возможности в дифференциации подвидов и более мелких таксономических единиц (популяций, экотипов) медоносной пчелы. С использованием мтДНК (Smith, 1991; Garneryetal., 1992), а в последующем микросателлитов (МС) (Estoup, 1995; Francketal., 2000) было подтверждено разделение A. mellifera на эволюционные ветви с некоторой перегруппировкой североафриканских подвидов между ветвями М и А.
Используя в качестве критерия индекс Fst[9], А.Estoup с соавторами (1995) дифференцировали африканские и западноевропейские подвиды с использованием семи МС. При этом степень дифференциации между подвидами внутри эволюционных ветвей была выше, чем между ними: Fst=0,34 и 0,25 соответственно. С использованием восьми МС было доказано гибридное происхождение пчел Пиренейского полуострова A. m. iberica (DeLaRuaetal., 2002), показано их родство с пчелами Балеарских островов Испании (DeLaRuaetal., 2003). F.Canovas с соавторами (2010), основываясь на анализе десяти МС, выявили тенденцию возрастания генетической гомогенности существующих экотипов у иберийских пчел (A. m. iberica) Пиренейского полуострова, обусловленную интенсивной практикой миграции, и возрастающего обмена семьями между пчеловодами. С использованием семи МС показана возможность дифференциации популяций трех подвидов A. mellifera, обитающих в России (Кривцов и др., 2011), дана оценка состояния аллелофонда и дифференциации внутрипородных линий пчел типа «Приокский» [1, 2].
Целью настоящей работы явилась характеристика генетической структуры и степени дифференциации популяций карпатских и краинских пчел с использованием микросателлитов.
Изучаемая выборка пчел была представлена популяциями A. m. carnica австрийского (CARN_AVS, 3 семьи, n=32) и польского (CARN_POL, 3 семьи, n=29) происхождения из коллекции ГНУ «НИИ пчеловодства Россельхозакадемии» ипопуляциями A. m. carpatica Майкопского типа (CARP_MAY, 8 семей, n=95), Закарпатья (CARP_ZAK, 5 семей, n=61) и Украины (CARP_UKR, 3 семьи, n=28), разводимыми в ФГУП «ППХ “Майкопский”». Для сравнительной оценки степени дифференциации краинской и карпатской пород в выборку были введены среднерусские (A. m. mellifera) (MR, 3 популяции, 13 семей, n=191) и серые горные кавказские пчелы (A. m. caucasica) (GMC, 7 семей, n=113).
Выделение ДНК проводили с использованием метода экстракции перхлоратом (Зиновьева и др., 1998). Мультиплексный анализ семи локусов МС (A024, A88, A113, AP043, HB-C16-05, HB-THE-03, HB-C16-01) выполняли на генетическом анализаторе АВI3130xl (AppliedBiosystems, США). Расчет популяционно-генетических параметров (среднее число и число эффективных аллелей на локус, степень наблюдаемой и ожидаемой гетерозиготности, показатели F-статистики) выполняли с использованием программного обеспечения GenAlEx (ver. 6.4). Генетическую структуру популяций медоносной пчелы на индивидуальном уровне оценивали с использованием коэффициента подобия Q[5]. Обсчет данных проводили с использованием программного обеспечения Structure (ver. 2.3.1) без введения предварительной информации о популяционной принадлежности особей для числа популяций (K-кластеров) от 2 до 4 с параметрами, описанными ранее [6]. Степень дифференциации популяций оценивали на основании значений Fst [9] и Rst(AMOVA) [8] при парном сравнении, а также генетических дистанций по M.Nei [4], средних значений коэффициента Q в соответствующих кластерах [5].
Результаты анализа общего числа аллелей и эффективных на локус показали несколько большее генетическое разнообразие карпатских пчел (12,4±2,7 против 10,0±2,1 и 4,5±1,3 против 3,8±0,6 соответственно), однако наблюдаемые различия были недостоверны. Число аллелей в отдельных локусах варьировало от 5 в A024 и до 26 в HB-C16-01 у карпатских и краинских пчел соответственно.
Анализ аллельных профилей показывает высокое генетическое сходство изучаемых пород пчел, на что указывает отсутствие достоверных различий в частотах встречаемости аллелей. Хотя в каждой из исследованных пород были обнаружены приватные аллели (25 в карпатской и 6 в краинской), все они встречались с относительно низкой частотой (от 0,3 до 2,0%), за исключением аллеля 192 локуса HB-THE-03, выявленного у карпатских пчел с частотой 9,2%.
 Расчет наблюдаемой степени гетерозиготности показал приблизительно равные значения данного показателя у карпатской и краинской пород: 0,58±0,042 и 0,57±0,037 соответственно, что было достоверно ниже ожидаемой степени гетерозиготности (–0,124 и –0,130 соответственно, p<0,05). На дефицит гетерозигот указывают также положительные значения индекса фиксации Fis: 0,16±0,052 и 0,17±0,064 соответственно. Анализ в аспекте отдельных популяций (табл. 1) показывает дефицит гетерозигот: от 5,2% в майкопской популяции карпатских пчел до 12,5% в австрийской популяции краинских пчел.
Расчет наблюдаемой степени гетерозиготности показал приблизительно равные значения данного показателя у карпатской и краинской пород: 0,58±0,042 и 0,57±0,037 соответственно, что было достоверно ниже ожидаемой степени гетерозиготности (–0,124 и –0,130 соответственно, p<0,05). На дефицит гетерозигот указывают также положительные значения индекса фиксации Fis: 0,16±0,052 и 0,17±0,064 соответственно. Анализ в аспекте отдельных популяций (табл. 1) показывает дефицит гетерозигот: от 5,2% в майкопской популяции карпатских пчел до 12,5% в австрийской популяции краинских пчел.
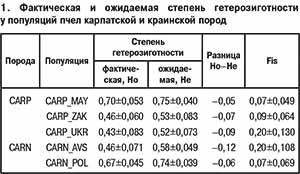
 Результаты анализа генетической структуры популяций при k=2 (рис. 1, а) показали наибольшую генетическую дифференциацию среднерусских пчел: Q2/2=0,86±0,016, в то время как значения членства в данном кластере для карпатской, краинской и серой горной кавказской пчел были существенно ниже и составили соответственно 0,06±0,009; 0,09±0,002 и 0,03±0,003. При k=3 (рис. 1, б) прослеживается четкая дифференциация изучаемых пород в соответствии с их принадлежностью к разным эволюционным ветвям: M (A. m. mellifera), C (A. m. carnica и A. m. carpatica), O (A. m. caucasica), значения членства в различных кластерах для которых составили соответственно Q1/3=0,80±0,018; Q2/3=0,65±0,026 и 0,85±0,022; Q3/3=0,96±0,002. При k=4, что соответствует числу исследуемых пород (рис. 1, в), сохраняется четкая дифференциация A. m. mellifera(Q2/4=0,78±0,021) и A. m. caucasica(Q3/4= 0,94±0,008), в то время как соответствующие значения у карпатских и краинских пчел составляют 0,03±0,049 и 0,05±0,099. Изучаемые популяции карпатской и краинской пород дифференцированы в меньшей степени, на что указывают близкие значения членства в общих кластерах: Q1/4=0,52±0,031 и 0,78±0,031; Q4/4=0,34±0,027 и 0,11±0,023 соответственно.
Результаты анализа генетической структуры популяций при k=2 (рис. 1, а) показали наибольшую генетическую дифференциацию среднерусских пчел: Q2/2=0,86±0,016, в то время как значения членства в данном кластере для карпатской, краинской и серой горной кавказской пчел были существенно ниже и составили соответственно 0,06±0,009; 0,09±0,002 и 0,03±0,003. При k=3 (рис. 1, б) прослеживается четкая дифференциация изучаемых пород в соответствии с их принадлежностью к разным эволюционным ветвям: M (A. m. mellifera), C (A. m. carnica и A. m. carpatica), O (A. m. caucasica), значения членства в различных кластерах для которых составили соответственно Q1/3=0,80±0,018; Q2/3=0,65±0,026 и 0,85±0,022; Q3/3=0,96±0,002. При k=4, что соответствует числу исследуемых пород (рис. 1, в), сохраняется четкая дифференциация A. m. mellifera(Q2/4=0,78±0,021) и A. m. caucasica(Q3/4= 0,94±0,008), в то время как соответствующие значения у карпатских и краинских пчел составляют 0,03±0,049 и 0,05±0,099. Изучаемые популяции карпатской и краинской пород дифференцированы в меньшей степени, на что указывают близкие значения членства в общих кластерах: Q1/4=0,52±0,031 и 0,78±0,031; Q4/4=0,34±0,027 и 0,11±0,023 соответственно.
 Результаты анализа генетической структуры A. m. carpatica и A. m. carnica в аспекте отдельных популяций представлены на рисунке 2. Эти данные подтверждают четкую дифференциацию карпатских и краинских пчел от среднерусских и серых горных кавказских. Высокая степень генетического сходства наблюдается между закарпатской и украинской популяциями карпатских и краинских пчел, на что указывают высокие значения членства в общем кластере (Q1/4=0,91±0,021; 0,82±0,057; 0,80±0,045 и 0,77±0,043). Майкопская популяция хотя и имеет некоторое генетическое сходство с другими карпатскими пчелами (Q1/4= 0,19±0,028), характеризуется иным происхождением, на что указывает относительно высокое значение членства в ином кластере: Q4/4=0,59±0,033 против (0,05±0,020) – (0,15±0,042).
Результаты анализа генетической структуры A. m. carpatica и A. m. carnica в аспекте отдельных популяций представлены на рисунке 2. Эти данные подтверждают четкую дифференциацию карпатских и краинских пчел от среднерусских и серых горных кавказских. Высокая степень генетического сходства наблюдается между закарпатской и украинской популяциями карпатских и краинских пчел, на что указывают высокие значения членства в общем кластере (Q1/4=0,91±0,021; 0,82±0,057; 0,80±0,045 и 0,77±0,043). Майкопская популяция хотя и имеет некоторое генетическое сходство с другими карпатскими пчелами (Q1/4= 0,19±0,028), характеризуется иным происхождением, на что указывает относительно высокое значение членства в ином кластере: Q4/4=0,59±0,033 против (0,05±0,020) – (0,15±0,042).
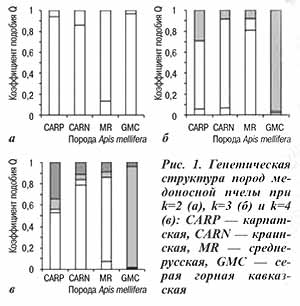
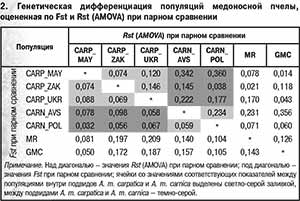 Результаты расчета Fst и Rst(AMOVA) при парном сравнении как меры генетической дифференциации изучаемых популяций приведены в таблице 2. При использовании в качестве критерия оценки значения Fst при парном сравнении наблюдается приблизительно равная степень дифференциации популяций карпатских и краинских пчел от среднерусских и серых горных кавказских. Карпатские пчелы в ряде случаев характеризуются более высокой степенью дифференциации друг с другом, чем с краинскими. Так, например, наблюдается большая дифференциация закарпатской популяции от майкопской и украинской популяций A. m. carpatica (Fst=0,074 и 0,069 соответственно), чем от польской A. m. carnica (Fst=0,056); украинская популяция в большей степени дифференцирована от майкопской и закарпатской популяций A. m. carpatica (Fst=0,088 и 0,069), чем от австрийской и польской популяций A. m. carnica (Fst=0,058 и 0,067 соответственно).
Результаты расчета Fst и Rst(AMOVA) при парном сравнении как меры генетической дифференциации изучаемых популяций приведены в таблице 2. При использовании в качестве критерия оценки значения Fst при парном сравнении наблюдается приблизительно равная степень дифференциации популяций карпатских и краинских пчел от среднерусских и серых горных кавказских. Карпатские пчелы в ряде случаев характеризуются более высокой степенью дифференциации друг с другом, чем с краинскими. Так, например, наблюдается большая дифференциация закарпатской популяции от майкопской и украинской популяций A. m. carpatica (Fst=0,074 и 0,069 соответственно), чем от польской A. m. carnica (Fst=0,056); украинская популяция в большей степени дифференцирована от майкопской и закарпатской популяций A. m. carpatica (Fst=0,088 и 0,069), чем от австрийской и польской популяций A. m. carnica (Fst=0,058 и 0,067 соответственно).
С другой стороны, доля межпопуляционной изменчивости между австрийской и польской популяциями A. m. carnica ниже, чем между майкопской A. m. carpaticaи польской A. m. carnica: 0,059 против 0,032. Использование в качестве критерия дифференциации популяций A. m. carnica и A. m. carpatica значений Rst(AMOVA) при парном сравнении в целом показывает сходные тенденции.
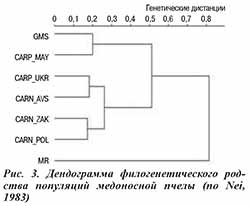 Генетические расстояния между исследуемыми популяциями пчел, рассчитанные по M.Nei (1983), представлены на рисунке 3. Результаты анализа генетических дистанций подтверждают данные об отличном происхождении майкопской популяции карпатских пчел. В отличие от других популяций A. m. carpatica и обеих A. m. carnica она характеризуется существенно большей близостью к серым горным кавказским пчелам (генетические дистанции 0,513 против 0,710–1,195 и 0,200 против 0,564–0,812 соответственно), что проявляется в формировании вышеназванными популяциями общей ветви на генеалогическом дереве (см. рис. 3). Полученные данные позволяют предполагать гибридное происхождение популяции майкопского типа.
Генетические расстояния между исследуемыми популяциями пчел, рассчитанные по M.Nei (1983), представлены на рисунке 3. Результаты анализа генетических дистанций подтверждают данные об отличном происхождении майкопской популяции карпатских пчел. В отличие от других популяций A. m. carpatica и обеих A. m. carnica она характеризуется существенно большей близостью к серым горным кавказским пчелам (генетические дистанции 0,513 против 0,710–1,195 и 0,200 против 0,564–0,812 соответственно), что проявляется в формировании вышеназванными популяциями общей ветви на генеалогическом дереве (см. рис. 3). Полученные данные позволяют предполагать гибридное происхождение популяции майкопского типа.
Таким образом, результаты проведенных нами исследований показывают, что микросателлиты являются хорошим инструментом дифференциации подвидов и популяций медоносной пчелы. Анализ аллельных профилей популяций карпатских и краинских пчел с использованием в качестве критериев оценки генетических различий значений Fst и Rst (AMOVA) при парном сравнении, а также генетических дистанций показывает, что степень дифференциации популяций внутри A. m. carpatica и A. m. carnica в ряде случаев выше, чем между A. m. carpatica и A. m. carnica, что можно рассматривать как указание на то, что A. m. carpatica является популяцией второго порядка A. m. carnica.
Результаты оценки генетических различий краинской и карпатской пород с использованием микросателлитов свидетельствуют об их общем происхождении и принадлежности к краинской породе (A. m. carnica), а карпатских — в качестве ее популяции.
Работа выполнена при финансовой поддержке Министерства образования и науки РФ, шифр 2012-1.4-12-000-1016-008.
В проведении исследований использовано оборудование ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ГНУ «ВИЖ Россельхозакадемии».
Н.А.ЗИНОВЬЕВА, М.С.ФОРНАРА, А.В.БОРОДАЧЕВ,
Е.А.ГЛАДЫРЬ, В.И.ЛЕБЕДЕВ, С.Н.АКИМОВА,
Н.И.КРИВЦОВ, Л.К.ЭРНСТ
ГНУ «НИИ пчеловодства Россельхозакадемии»
Ключевые слова:
ДНК-анализ, популляции, микросателлиты, краинская порода, карпатская порода.
Аннотация:
проведенные исследования показали, что микросателлиты являются хорошим инструментом дифференциации подвидов и популяций медоносной пчелы, а также результаты оценки генетических различий краинской и карпатской пород с использованием микросателлитов свидетельствуют об их общем происхождении и принадлежности к краинской породе (A. m. carnica), а карпатских — в качестве ее популяции.
Литература:
1. Зиновьева Н.А., Кривцов Н.И., Форнара М.С., Бородачев А.В., Бородачев В.А., Березин А.С., Гладырь Е.А. Оценка изменчивости аллелофонда микросателлитов при создании специализированных линий медоносной пчелы среднерусской породы // Достижения науки и техники АПК. — 2011. — № 10.
2. Зиновьева Н.А., Кривцов Н.И., Форнара М.С., Гладырь Е.А., Бородачев А.В., Лебедев В.И. Микросателлиты как инструмент оценки динамики аллелофонда при создании типа «Приокский» среднерусской породы медоносной пчелы // Сельскохозяйственная биология. — 2011. — № 6.
3. Кривцов Н.И., Горячева И.И. Генетический анализ внутривидовой структуры пчелы медоносной // Пчеловодство. — 2009. — № 10.
4. Nei M., Tajima F., Tateno, Y. Accuracy of estimated phylogenetic trees from molecular data // J. Mol. Evol. — 1983. — 19.
5. Pritchard J. K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data // Genetics. — 2000. — 155.
6. Raffaele D’O., Alberto M., Marco L., Robin F.A.M. Genetic characterization of Italian honeybees, Apis mellifera ligustica, based on microsatellite DNA polymorphisms // Apidologie. 2007. № 38.
7. Ruttner F. Biogeography and Taxonomy of Honeybees. Springer Verlag. — Berlin, 1988.
8. Slatkin M. A measure of population subdivision based on microsatellite allele frequencies // Genetics, 1995, 139, 1463.
9. Weir B.S., Cockerham C.C. Estimating F-statistics for the analysis of population structure // Evolution. 1984. — №38.

Ранние матки — спокойствие на 6 сотках…
янв 29, 2019
Получаем высококачественный и экологичес…
июнь 13, 2020
Сезонные изменения физиологического сост…
июль 21, 2022
Сотовый мед в тарелочке…
дек 25, 2014
В чем изъян ульев XX века и чем отличает…
авг 10, 2016
Технология двухсемейного содержания пчел…
окт 21, 2022
Искусственные соты для вывода маток…
сен 6, 2016
Кормление пчел сахаром…
июль 27, 2014
Влияние временных факторов на содержание…
март 19, 2025
Жить человеку в мире с пчелой…
нояб 4, 2016
Кормушка-поилка
апр 25, 2023
Использование роевых семей (2)…
мая 17, 2015















 Адрес редакции журнала "Пчеловодство":
Адрес редакции журнала "Пчеловодство":